3D-QSAR也可以很酷!
原文来自:http://www.cresset-group.com/wp-content/uploads/2012/09/3D-QSAR-Why-be-square_FINAL.pdf
作 者:M Mackey, T Cheeseright,J Melville, R Scoffin, C Earnshaw
本文介绍了Forge进行3D-QSAR的基本原理与优势。总的来说,Forge集成了活性构象建模,分子叠合,3D-QSAR建模(PLS)一整套流程。这个流程基本可以自动执行,您不必再花几周、几个月时间去试各种叠合,现在几分钟就可以完成传统3D-QSAR枯燥的工作。
前言
基于分子相互作用势的3D-QSAR是一种由来已久的技术。然而,目前的技术存在着许多问题,使得可靠地产生有效模型变得困难。这些问题包括:
- 叠合问题(作为计算描述符的前提条件,如何以3D形式叠合分子);
- 采样问题(正如广泛应用的,在网格上采样产生边缘效应和额外的噪音);
- 描述符问题(分子性质在相同的采样位点上计算,这些性质的准确度如何?)
Cresset发布了ForgeV10,一种生成3D-QSAR模型的全新的集成方法,它使用我们的“场点(field point)”描述符。这些描述符首先用于叠合分子,要么使用自由叠合或者先叠合公共子结构。一旦分子叠好后,训练集的场点集合将用于导出一组规范不变的采样点,以便在确保分子周围的可能对活性有贡献的区域获得充足的采样的同时,减少需要考虑的描述符的数量。最后,计算采用值与构建偏最小二乘(Partial Least Square, PLS)模型。
场

通过定位静电和立体场局部最大的空间中的点产生分子的场点。每一个这样的点都在点上用场强标记。我们使用四中场:正的和负的静电场、“形状场”(范德华力van der Waals)和“疏水场”(一个与立体位阻和疏水性相关的密度函数)。
深入处理XED力场提供的静电势对于获得良好的分子静电势是必须的。
场的采样
给定一组已经叠合好的分子,他们聚集的场点定义了空间中的一组采样点。我们采用球排除算法来过滤这组采样点,然后为每个分子计算场值,产生数据矩阵,进而使用PLS。这种技术类似于CoMFActor,但不是在分子周围的格点上采样,我们进对一个活多个分子存在场点的地方采样,这是通过构建得到的最重要的地方。相对CoMFA的优势是您能获得许多比基于网格的采样更少的采样点,不存在规范变化问题,采样位点是物理选择而非任意选择,并且有一个分子叠合与QSAR一致的框架。
回溯性测试
针对文献CoMFA数据集进行测试:
- 15个叠合可用的数据集。
- CoMFA的平均交叉验证RMSE是0.72。
- 使用CoMFA叠合的场QSAR是0.74。
- 简单模型(体积指示变量——1代表里边,0代表外面)是0.83。
通过大量广泛的数据集,我们发现体积指标变量在模型性能与解释能力方面均一致优于vdW势能。增加一个疏水势一般不能改善模型。
来自NK3的例子
- 应用forgeV10/FieldTemplate模拟生物活性构象。
- 将我们的互变异构体进行分类。
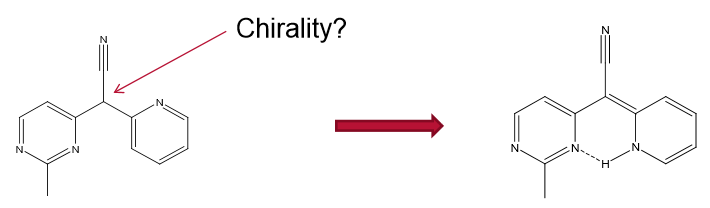
- 叠合所有分子到模板上,并计算描述符。
- 应用PLS。
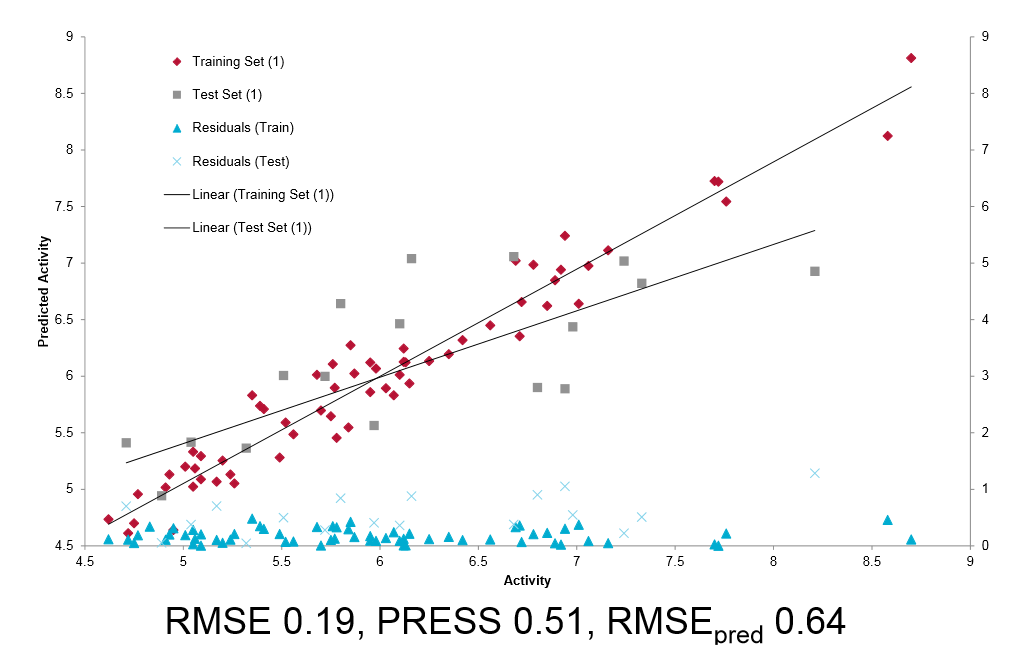
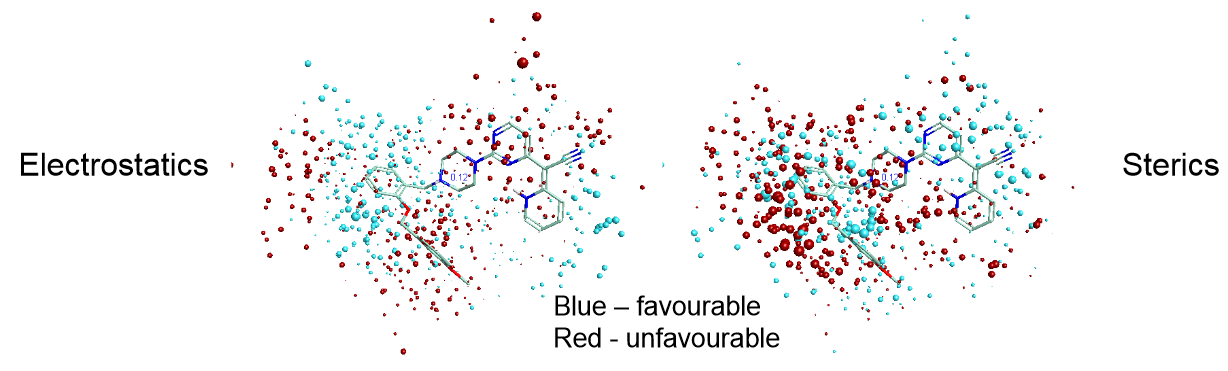
- 对不同系列重复上述步骤。
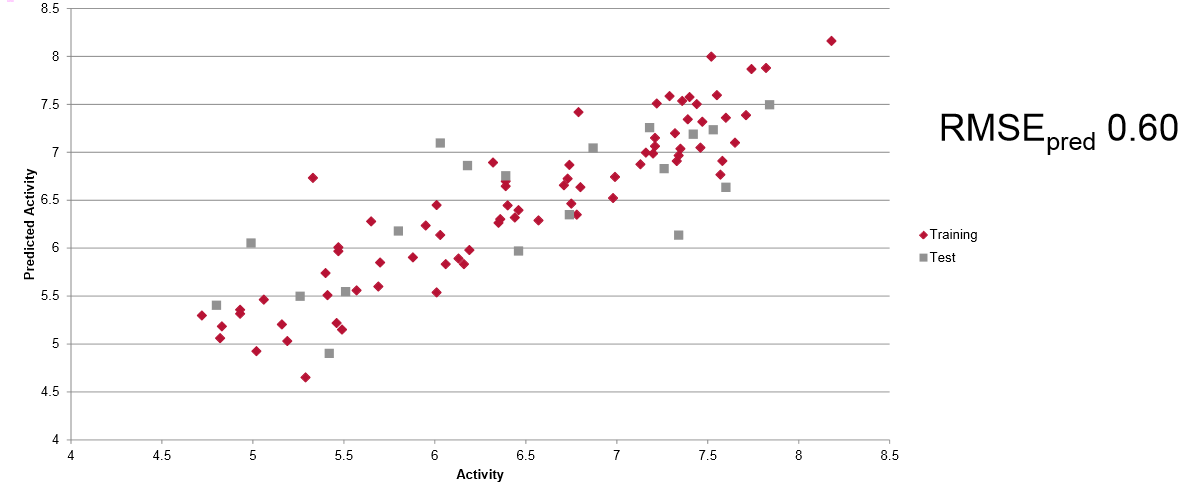
总结
使用分子场采样的QSAR相对于基于网格的采用方法有着重大优势。网格大小或间隔不需要实行参数化。这个方法是规范不变的和可重现的,由于采样位点放置在建模空间中最重要的区域,边缘效应得到降低。
采用该技术的新的软件,forgeV10,现在可从Cresset获得。
参考文献
1 Cheeseright, T.; Mackey, M.; Rose, S.; Vinter, A. Molecular Field Extrema as Descriptors of Biological Activity: Definition and Validation. J. Chem. Inf. Model. 2006, 46, 665-676.
